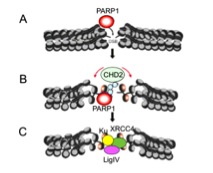
図1 DNA修復の分子機構
二本鎖切断の際の非相同末端結合(NHEJ)による修復機構を例として示す。まず,PARP1が損傷を認識し,最初に損傷DNA部位に集積する(A)。PARP1に続いてクロマチン再構成因子CHD2(B),さらにDNA修復因子群(C)がリクルートされることで,最終的にDNA損傷が修復される。(Luijsterburg et al, Mol. Cellを改変)
DNA修復の仕組みを解明
〜乳がん治療への希望〜
【ポイント】
* 細胞のDNA修復の基盤となる新しいタンパク質複合体を発見した
* 新規タンパク質複合体の阻害は、乳がんの腫瘍形成を抑制する
【概要】
わたしたちのからだの細胞の遺伝情報であるDNAはたえず様々な損傷を受けると同時に,いくつかの修復機構によって安定に保たれています。しかし,DNAが不安定化してその異常が蓄積するとがんを発症します。そして,その後のがんの進展はDNA修復機構の働きに支えられています。特に,BRCA1遺伝子の変異をもつ乳がんはDNA修復機構に強く依存していることが知られています。中井彰教授らの研究グループは,DNA修復に必要な新しいタンパク質複合体を世界で初めて発見しました。さらに,この複合体ができない条件下では,マウスでのBRCA1変異乳がん細胞の腫瘍形成が顕著に抑制されることも明らかにしました。したがって,この複合体の発見は,がんの治療薬の開発に結びつく可能性があります。
【背景】
わたしたちの細胞は,遺伝情報を担う染色体のDNAやその指令を実行するタンパク質などで出来ており,それらのバランスが維持されることで健康が保たれています。これらDNAやタンパク質の異常をひき起す環境ストレス(紫外線や温度変化など)や代謝によるストレス(活性酸素など)に対処するために,細胞は優れた適応機構を備えています。その一つがDNA損傷に対するDNA修復機構(注1)です。
DNA損傷には紫外線などによる一本鎖DNA損傷と,放射線や抗がん剤などによる二本鎖DNA損傷があります。いずれの場合にも,最初に損傷DNA部位に集積するのが損傷を認識するポリ(ADP-リボース)合成酵素1(PARP1)(注2)です。PARP1に続いて多くのDNA修復酵素が集積することで,最終的にDNA損傷が修復されます(図1)。つまり,PARP1はDNA修復の鍵因子と言えます。しかし,PARP1がどこから来てDNA損傷部位へ再分布するのか不明でした。
一方、細胞にはタンパク質の異常に対処するための適応機構も備えており、進化的にも保存されたものの一つが熱ショック応答と呼ばれる転写誘導機構です。それを調節する鍵因子が熱ショック転写因子1(HSF1)(注3)です。HSF1は温熱ストレス条件下などでタンパク質の異常が生じると,それを修復する遺伝子の上流に結合して転写を誘導します。最近,HSF1があらかじめ染色体DNAのあらゆる部位に結合することが示されてきましたが(図2),その意義は十分には解明されていませんでした。
【研究の成果】
研究グループは,以前の研究でヒトHSF1と相互作用するタンパク質群を網羅的に同定しており,その中でPARPファミリー(17因子からなる)との複合体の役割の解明に焦点を絞って研究を進めました。まず,HSF1-PARP13-PARP1の新しい三者複合体を同定し,PARP13が足場タンパク質としてHSF1とPARP1とに直接結合することを見出しました(図3)。次に,ChIP-seq法(注4)などによって,この三者複合体がHSF1を介して染色体の全体にわたり結合していることを示唆しました(図4)。
染色体上に存在する三者複合体機能の解明の糸口となるのが,それが調節する遺伝子群です。研究グループは,DNAマイクロアレイ法(注5)を用いた網羅的遺伝子発現解析により,三者複合体がDNA損傷の際に誘導される遺伝子群の発現制御に関与することを見出しました(詳細は省略、原著論文を参照)。PARP1がDNA修復の過程に直接関与することも合わせると,三者複合体がDNA修復に重要な役割を担うことが示唆されました。実際に,内在性のHSF1を複合体形成ができない変異体HSF1-A20Aに置換すると,DNA損傷薬剤(ドキソルビシン)によるDNA修復因子のDNAへの集積は顕著に減少し,DNA損傷がより蓄積しました(図5)。さらに,二本鎖DNA損傷の2つの修復機構(相同組換え修復,非相同末端結合)をモニターできる実験系を用いることで,三者複合体が染色体上に存在することがPARP1の損傷部位への集積を促進し,相同組換え修復の効率を高めることも明らかとなりました(図6)。
がんは,DNA損傷の増加あるいはDNA修復機能の低下によって,DNA異常が蓄積することで発症します。細胞ががん化するとDNAがより不安定化するために,がんの進展はDNA修復機構の働きに支えられます。DNA修復因子の一つがBRCA1で、BRCA1遺伝子(注6)に変異をもつ乳がんの進展はほとんどのDNA修復機構に関与するPARP1活性に強く依存していることが知られています。研究グループは,三者複合体形成の阻害はBRCA1遺伝子に点変異を持つヒト乳がん細胞の増殖を低下させることを見出しました。さらに,三者複合体を阻害するとBRCA1遺伝子を欠損するマウス乳がん細胞の増殖,およびマウスでの腫瘍形成が顕著に抑制されることを明らかにしました(図7)。
【研究の意義と今後の展望】
細胞が正しく機能するためにDNAの恒常性を維持することは必要であり,そのためにDNA損傷の種類に対応して複数のDNA修復機構が存在します。いずれの機構においてもPARP1は必須の因子であり,DNA損傷の際にPARP1 がDNA損傷部位へ再分布することが必須であることが知られています。しかし,このPARP1がどこから来るのかを含めて,その再分布の機構については不明でした。本研究により,染色体DNAに結合するHSF1がPARP13を介してPARP1をあらかじめ全染色体上に留めておくことが,PARP1のDNA損傷部位への集積に必要であることが明らかとなりました(図8)。PARP1を速やかにDNA損傷部位へ集めるために,太古の昔から存在するDNA結合因子HSF1を利用するという生物の適応戦略が解明されました。
乳がんは女性のがんで最も罹患率が高く,死因としても上位に位置します。乳がんと関連する遺伝子としてBRCA1遺伝子は最も良く知られています。近年,BRCA1遺伝子変異を持つ乳がん,卵巣がん,膵臓がん,前立腺がんなどの治療ターゲットとしてPARP1 が注目されており,PARP1阻害剤の臨床試験が世界中で進行しています。しかし,PARP1は染色体構造,転写,DNA修復,様々なシグナルなどを調節する多機能タンパク質であることから副作用も懸念されます。本研究により,PARP1の酵素活性を残したままでHSF1-PARP13-PARP1複合体形成を阻害することにより,BRCA1遺伝子変異を持つ乳がんの進展を抑制できることが分りました。つまり,HSF1-PARP13-PARP1の相互作用を断ち切る化合物を見いだすことで、この三者複合体がより副作用のないがんの治療ターゲットとなりうる可能性を示しました。
<論文情報>
Fujimoto M, Takii R, Takaki E, Katiyar A, Nakato R, Shirahige K, and Nakai A. The HSF1-PARP13-PARP1 complex facilitates DNA repair and promotes mammary tumorigenesis. Nature Communications 8, 1638, 2017.
doi: 10.1038/s41467-017-01807-7.
<問い合わせ先>
国立大学法人山口大学大学院医学系研究科医化学講座
教授 中井 彰
TEL: 0836-22-2214 FAX: 0836-22-2315
<補足説明>
(注1)DNA修復機構
DNAの様々な損傷を修復する過程で,一本鎖DNA損傷と二本鎖DNA損傷の修復に対して,それぞれいくつかの機構が知られている。前者に対する3つの修復機構の解明に対して2015年にノーベル賞が授与された。後者に対しては相同組換え修復(HRR)と非相同末端結合(NHEJ)の2つの修復機構が知られている。PARP1はほとんどの修復機構に関与する。
(注2)ポリ(ADP-リボース)合成酵素1(PARP1)
PARPは,自分自身を含むタンパク質にADP-リボースを付加する修飾酵素群である。PARP1をはじめとする17種のPARPファミリー分子がある。PARP13に酵素活性はない。DNA 修復,転写調節,染色体構造の調節などの多くの機能に関与する。PARP1阻害剤は,BRCA1遺伝子変異のあるがん細胞の増殖を顕著に抑制する。BRCA1遺伝子変異を持つ乳がん,卵巣がん,膵臓がんなどに対して,多種類のPARP1阻害剤の臨床試験が世界中で進められている。
(注3)熱ショック転写因子1(HSF1)
ある生育温度で維持されている細胞を数度高い温度にさらすと,タンパク質の異常を修復する熱ショックタンパク質が誘導される。この応答は熱ショック応答とよばれ,これを転写(DNAからRNAへの読み取り)のレベルで調節するのがDNAに結合する熱ショック転写因子1(HSF1)である。がん細胞はタンパク質異常を起こしやすく,その増殖はHSF1にも依存することが知られている。
(注4)ChIP-seq法
クロマチン免疫沈降シークエンス法の略称。転写因子(DNA結合タンパク質)などの染色体上での結合部位を,網羅的に解析できる。免疫沈降で回収したDNA断片を,超高速シークエンシングを行うことで半定量化する。
(注5)DNAマイクロアレイ法
細胞の持つ全ての遺伝子の発現をRNAレベルで定量的に計測する方法である。スライドガラス上に数万個のDNA断片をのせ,細胞から抽出したRNAとハイブリッドを形成させ,ハイブリッド形成の強度を指標にして各遺伝子のRNA量を測定する。
(注6)BRCA1遺伝子
がん抑制遺伝子の一つで,DNAの相同組換え修復(HRR)に関与する。その変異により遺伝子不安定性を生じ,最終的に乳癌やがんをひき起こす。ハリウッド女優のアンジェリーナ・ジョリーさんはこの遺伝子に変異を持ち,予防的処置として両方の乳房を切除したことで知られる。
<図と説明>

図1 DNA修復の分子機構
二本鎖切断の際の非相同末端結合(NHEJ)による修復機構を例として示す。まず,PARP1が損傷を認識し,最初に損傷DNA部位に集積する(A)。PARP1に続いてクロマチン再構成因子CHD2(B),さらにDNA修復因子群(C)がリクルートされることで,最終的にDNA損傷が修復される。(Luijsterburg et al, Mol. Cellを改変)

図2 HSF1の染色体への結合部位
(A)ChIP-seq法を用いて,マウス胎児繊維芽細胞におけるHSF1の染色体結合部位を示す。通常条件(37℃)では316のHSF1結合部位,熱ストレス条件下(42℃,30分)では2,291のHSF1結合部位が同定されている。(B)3つの熱ストレス条件下で転写誘導される遺伝子周辺のHSF1結合部位のピークを示す。特に,矢印は通常条件下でのピークを示す。(Takii et al, Mol. Cell. Biol. 2015を改変)

図3 HSF1,PARP1,PARP13の直接的な相互作用の解析
(A)PARP13,GSTと融合したPARP1,GSTと融合したHSF1を精製し,GST-pull down 法により直接的な相互作用を調べた。PARP13は精製GST-PARP1および精製GST-HSF1とともに沈降する。(B)三者複合体の模式図。HSF1は三量体を形成してDNAへ結合出来るが,同時にPARP13を介してPARP1とも相互作用する。
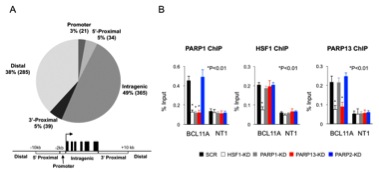
図4 PARP1の染色体への結合部位の同定
(A)ChIP-seq法によりヒトHeLa細胞における744のPARP1結合部位を同定し,それらが遺伝子のどの領域かを示した。PARP13を欠くと,この中でわずか10のPARP1結合部位が残る(データは示さない)。(B)BCL11A遺伝子座に焦点を当てて,PARP1、HSF1、PARP13の結合をChIP法で調べた。HSF1あるいはPARP13をノックダウン(HSF1-KD, PARP13-KD)するとPARP1の結合が失われる。以上の結果は,三者複合体がHSF1を介して染色体の全体にわたり結合していることを示唆する。星印はt検定によって有意差があることを示す。
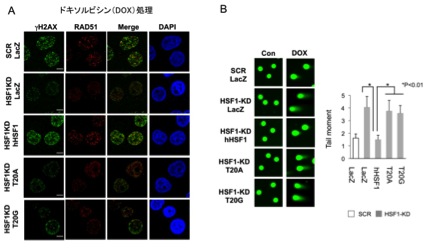
図5 三者複合体がDNA修復因子の集積を促進する
(A)細胞をDNA損傷薬剤(ドキソルビシン,DOX)で処理すると修復シグナルであるγH2AX染色が増強し,修復因子RAD51が染色体へ集積する(1列目)。 HSF1を欠損するとそれらが減弱する(2列目)。そこで,野生型HSF1を戻すとγH2AX染色の増強,修復因子RAD51の集積を認めるが(3列目),変異型HSF1(T20A, T20G)を戻しても認めない(4,5列目)。(B)コメットアッセイ法によってDNA損傷の程度を調べた。細胞にドキソルビシン(DOX)を処理するとDNA損傷が蓄積するが,HSF1欠損(HSF1-KD)によってより顕著となる。野生型HSF1(HSF1)を発現させるとDNA損傷はもとのレベルまで低下するが,変異体HSF1(H20A, T20G)を発現させても高いままである。

図6 三者複合体がPARP1の損傷部位への集積を促進する
(A)二本鎖DNA損傷の際の相同組換え修復(HRR)をモニターするためのレポーター遺伝子の構造を示す。SceGFPとiGFPはともに蛍光タンパク質GFPの一部を持つ不完全なものである。制限酵素I-SceIでSceGFP遺伝子の一部を切断すると,HRRにより完全なGFPが出来る。(B)PARP1の切断部位近傍(SCE-1)への集積をChIPアッセイ法で調べた。変異HSF1(T20A)を発現する細胞では集積が減少した(赤)。(C)相同組換え修復の効率をGFPの蛍光を発する細胞の数として定量した。変異HSF1(T20A)を発現する細胞ではHRRの効率が低い。

図7 BRCA1遺伝子を欠損するマウス乳がん細胞の腫瘍形成
(A)BRCA1遺伝子を欠損するマウス乳がん細胞(KB1P-G3, KB1P-B11)の細胞増殖を調べた。HSF1を欠損する(HSF1KD),あるいは三者複合体を形成できない変異HSF1(T20A, T20G)を発現するがん細胞の増殖は低下した。星印はANOVA検定によって有意差があることを示す。(B)KB1P-G3細胞のマウス乳腺近傍での腫瘍形成を生体イメージングシステム(IVIS)調べた。三者複合体を形成できない変異HSF1(T20A, T20G)を発現する細胞のマウスでの腫瘍形成は顕著に低下した。
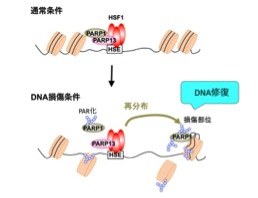
図8 PARP1の染色体への結合とDNA損傷に伴う再分布
通常条件下では,染色体DNAに結合するHSF1がPARP13を介してPARP1をあらかじめ全染色体上に留めておく。DNA損傷によりPARP1がPAR化活性を獲得し,自らをPARP化することで解離し,DNA損傷部位へ再分布する。あらかじめ三者複合体を作ることが,PARP1のDNA損傷部位への集積に必要である。PARP1は、DNA修復因子群をひき寄せることで修復を促進する。